Modellierung dynamischer Prozesse mit Differentialgleichungen (DGLs) und deren Parameterschätzung mit dem Excel® Solver
Demonstrationsbeispiel: Verläufe von Epidemien/Pandemien
Zusammenfassung: Dieses Projekt erarbeitet eine Möglichkeit, wie man in Excel® mit Hilfe des implementierten Solver die Parameterschätzung von numerisch gelösten Differentialgleichungen, bzw. -systemen, erfolgreich durchführen kann. Dazu werden Beispiele mit unterschiedlicher Komplexität vorgestellt.
SEIR, SEIRD, SEIRD_HI Modelle
Aus gegebenen Anlass der aktuellen Covid 19-Dynamik (Stand Januar/April 2022) wird ein epidemiologisches Stamdardmodell zur Ausbreitung von Krankheiten (z.B. Virusinfektion) als weiteres Bespiel für die Parameterschätzung mit dem Solver hinzugefügt, von der erweiterten Komplexität her die logische Fortsetzung des pharmakokinetischen Beispiels der Einleitung des Differentialgleichungprojekts:
Zur besseren Erläuterung der Hintergründe und Limitierungen, bzw. zahlreichen Annahmen von solchen Epidemiemodellen verweisen wir auf die Wikepedia Seite für SEIR Modelle.
Kurz zusammengefasst: eine Population von N Individuen sei zerlegt in die vier Kompartimente S, E, I, R, d. h. die Population N wird als Gesamtheit betrachtet. Jedes Individuum der Population N kann theoretisch die Prozedur Susceptible (S) → Exposed (E) → Infectious (I) → Recovered (R) (und optional Death (D)) durchlaufen.Die Ausbreitungsdynamik der Krankheit wird beschrieben durch ein System von Differentialgleichungen mit den folgenden Parameter:
- β:Transmissionsrate der spezifischen Krankheit, besteht prinzipiell aus dem Produkt zweier Parameter, der Transmissionsrate Tr, welche tatsächlich Virus,- bzw. Variantenspezisch ist, und der Kontaktrate Kr, welcher sozusagen das Verhalten der Population N kondensiert. Tatsächlich wird in der Grundannahme β als zeitkonstant angenommen, realistisch wäre eine zeitabhängige Funktion. Im Folgenden werden beide Parameter zeitkonstant ins Modell übernommen.
- α: der Übergangsrate
- γ: der Erholungsrate
- pd: der Sterberate
Die zahlreichen weiteren Modellannahmen der SEIR, SEIRD Modelle werden hier nicht wiederholt, es wird nochmals auf die Wikepedia Seite für SEIR Modelle verwiesen. Es ergibt sich folgendes Modell:
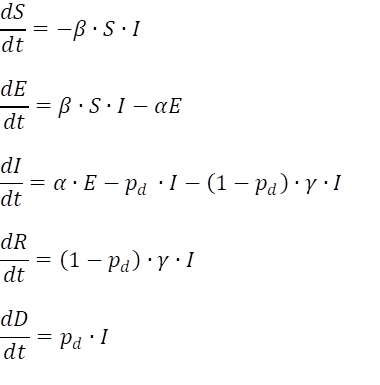
mit folgendem VB Code für das Gleichungssystem:
Sub ODE(y#(), dy#())
dy(1) = -(Kr * Tr) * y(1) * y(3) # mit der Kontaktrate Kr und der Transmissionsrate Tr
dy(2) = (Kr * Tr) * y(1) * y(3) - alpha * y(2)
dy(3) = alpha * y(2) - pd * y(3) - (1 - pd) * gamma * y(3)
dy(4) = (1 - pd) * gamma * y(3)
dy(5) = pd * y(3)
Modellbeschreibung:
Aus der Gesamtpopulation N sind zum Zeitpunkt t=0 gewisse Anteile an Anfälligen und Infizierten vorhanden. Mit der Transmissionsrate β (= kb) gehen diese in
Gruppe der Exponierten über, die wiederum mit der Rate α in die Gruppe der Infizierten übergehen. Die Infizierten reduzieren sich mit der
Sterbewahrscheinlichkeit pd, die dann im Kompartiment D auftauchen oder gehen mit der Gegenwahrscheinlichkeit (1 - pd) und der
Erholungsrate γ in das Kompartiment der Genesenen über (R), die im Idealfall dann immun gegen das Virus sind.
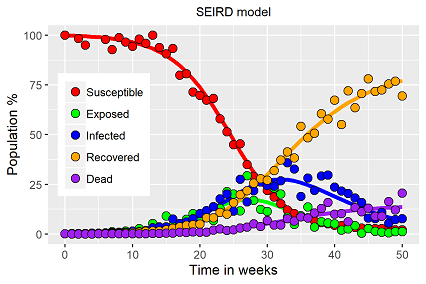
Anhand des Modellbeispiels wurde ein künstlicher Datensatz generiert, an dem über die VBA basierten Solverfunktionen die Modellparameter geschätzt werden können.
| No | Eo | Io | Kr | Tr | α | γ | pd |
| 99.77 | 0 | 0.23 | 0.799 | 0.799 | 0.294 | 0.123 | 0.022 |
Mit Hilfe der simultanen Parameterschätzung lässt sich anhand der Parameterwerte das Virus im spezifischen Rahmen, z.B. der genetischen Exposition, allgemeiner Gesundheitszustand, Widerstandsfähigkeit und/oder Altersstruktur der Population N spezifizieren. Von Interesse ist dabei der Parametervektor von Tr bis pd, der das eigentliche Virusverhalten beschreibt, natürlich immer im Zusammenhang zum Resistenzgrad der zugrundliegenden Population. Änderungen in den Parametern deuten dann auf Änderungen der Virusvariante, bzw. Änderung der Immunität hin. Gleichzeitig beschreibt Kr, die Kontaktrate, das Verhalten der Population. Es ist zu erwarten, dass dieser Parameter ebenfalls mit der Zeit variiert. Vielleicht beschreibt der Begriff "Sorglosfaktor" den Parameter Kr empirisch gesehen weitaus präziser. Zu Beginn der Virusausbreitung gehen die potentiell Anfälligen (also die Gesamtpopulation) erfahrungsgemäß sorglos mit einer Pandemie um, erst wenn die "Gefahr" vermeintlich näherrückt, verkleinert sich die potentielle Kontaktrate, entweder durch eigenverantwortliche Maßnahmen oder durch staatliche Restriktionen.
Erweitertes Beispiel
Gerade auch im Hinblick auf das strukturelle Problem von Hospitalisierung und Intensivstation und den damit verbunden Sterberaten der aktuellen Pandemie kann das Modell entsprechend erweitert werden. Es werden die weiteren Kompartimente (und Gleichungen) für die Modellierung der Hospitalisierung und Weiterleitung auf die Intensivstationen erweitert. Die übrigen Differentialgleichungen werden um die neuen Raten und Gegenwahrscheinlichkeiten erweitert, vor allem um die Erholungs- und Sterberaten aus jedem Kompartiment. Das folgende Gleichungssystem ist durch die zwei Extrakompartimente wesentlich komplexer:
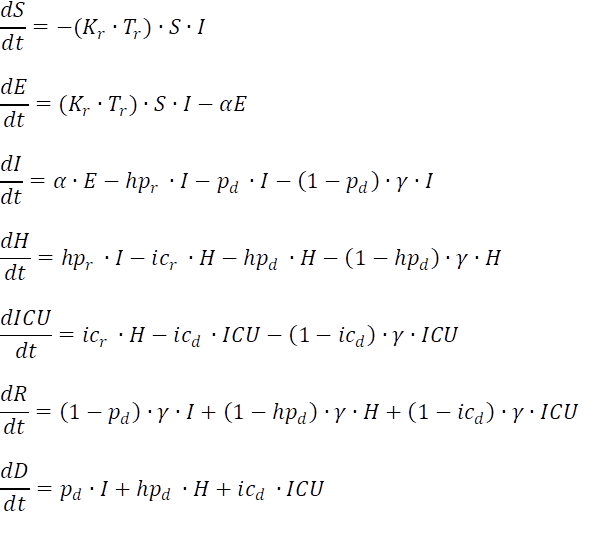
wiederum mit folgendem VB Code für das Gleichungssystem:
Sub ODE(y#(), dy#())dy(1) = -(Kr * Tr) * y(1) * y(3)
dy(2) = (Kr * Tr) * y(1) * y(3) - alpha * y(2)
dy(3) = alpha * y(2) - hpr * y(3) - pd * y(3) - (1 - pd) * gamma * y(3)
dy(4) = hpr * y(3) - ir * y(4) - hdr * y(4) - (1 - hdr) * gamma * y(4)
dy(5) = ir * y(4) - idr * y(5) - (1 - idr) * gamma * y(5)
dy(6) = (1 - rd) * gamma * y(3) + (1 - hdr) * gamma * y(4) + (1 - idr) * gamma * y(5)
dy(7) = pd * y(3) + hdr * y(4) + idr * y(5)
Fortsetzung der Modellbeschreibung ab dem Stadium der Infizierten:
Aus der Gruppe der Infizierten bestehen jetzt drei Abflüsse, zusätzlich zu den Genesenden und direkt Verstorbenen kommen die Hospitalisierten, die
dann mit der entsprechenden Rate im Kompartiment H auftauchen. Die Hospitalisierten reduzieren sich, zusätzlich zu den schon erwähnten Wahrscheinlichkeiten,
um die Patienten, die auf die Intensivstation (ICU) mit der Rate icr weitergereicht werden. Das Kompartiment der Intensivstation entleert sich dann wieder
mit der Sterberate icd, bzw. der Genesungsrate. Um das System nicht
zusätzlich zu komplizieren wird eine allgemeine Genesungsrate für alle
entsprechenden Kompartimente angenommen. DIe Genesenden und Toten werden dann jeweils wieder alle aus den vorherigen drei Kompartimenten gesammelt.
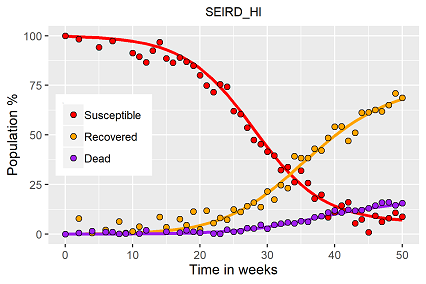
Basierend auf den Modellannahmen wurde ebenfalls ein künstlicher Datensatz generiert, an dem über die VBA basierten Solverfunktionen die Modellparameter geschätzt werden können.
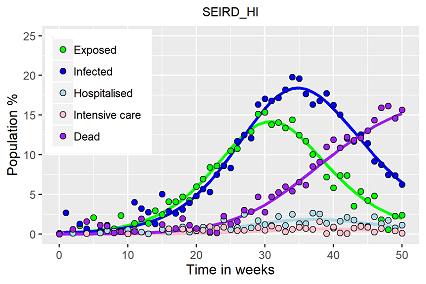
| No | Eo | Io | Kr | Tr | α | γ | pd | hpr | icr | hdr | idr |
| 99.76 | 0 | 0.24 | 0.801 | 0.801 | 0.302 | 0.149 | 0.0001 | 0.057 | 0.377 | 0 | 0.992 |
Auch dieses komplexere Modell lässt sich gut mit dem Solver anpassen, zeigt aber auch potentielle Schwächen. Anhand des Fits ergibt sich eine extrem hohe Sterberate, wenn man das Stadium "Intensivstation (ICU) erreicht hat, ein bisschen wie es in der 1. Welle der Pandemie zu beobachten war. Man muss hier zugeben, die zunehmende Modellkomplexität führt zu mehreren Lösungen, zwischen denen anhand der bestehenden Daten nicht unterschieden werden kann.
Bezugnehmend auf die aktuelle Pandemie: um überhaupt einen proportional graphischen Effekt mit den SEIR Modellen in den beschriebenen Kompartimenten darstellen zu können, mussten die Parameter so hoch gewählt werden, dass das Modell über die Simulation eines Jahres mit einer Welle und konstanten Parametern bei 95% Infizierten, 75% Genesenen und 15% Toten der Population N endet. Es ist also mehr als offensichtlich, so ist es in Deutschland (und auch sonst nirgendwo) nicht verlaufen, selbst nach über 2 Jahren liegt die Infizierten/Genesenen bei ca. 25%, die Sterberate auf die Gesamtbevölkerung gesehen bei durchschnittlich 0.17%.
 Evaluation reinvented
Evaluation reinvented
