Details der hyperspektralen Bildanalyse für den interessierten Leser
Wiederholung:  Die im Nemaplot Projekt entwickelte Technik ist ebenfalls auf die Analyse hyperspektraler Bilder
anwendbar. Auch hier werden die
Gesamtinformationen des Spektrums, diesmal für jedes Pixel eines Bildes, durch die Modellanpassung berücksichtigt.
Im Gegensatz zu den Experimenten mit einer durch den Versuch bedingten Klasseneinteilung sind diese Klasseneinteilungen
für eine Analyse innerhalb des Bildes nicht möglich, da hier in einem Bild alle möglichen Facetten eines Einflussfaktors dargestellt werden. Daher werden die
Analysen des zweiten Auswerteschritts mit einer Clusternalyse durchgeführt. Der Vorteil der Clusteranalyse besteht darin, das keine Klassifikation vorgegeben
werden muss.
Als Beispiel wird das Falschem Mehltau, (Plasmopara viticola))
Versuchen gewählt1. Parallel zu den Blattmessungen mit der Plant
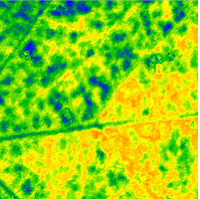
Probe wurden ganze Blätter mit einer hyperspektralen Linienkamera gescannt. Das RGB
Bild (rechts) ist ein Ausschnitt der Oberseite eines Blattes. Die Frage gilt zu klären
was, welche pathogen bedingten Muster der Blattunterseite durch die
Modellanpassung auf der Blattoberseite erkannt werden können. Dazu wird das Problem nach der Modellanpassung in mehrere Teilschritte
aufgeteilt.
Die im Nemaplot Projekt entwickelte Technik ist ebenfalls auf die Analyse hyperspektraler Bilder
anwendbar. Auch hier werden die
Gesamtinformationen des Spektrums, diesmal für jedes Pixel eines Bildes, durch die Modellanpassung berücksichtigt.
Im Gegensatz zu den Experimenten mit einer durch den Versuch bedingten Klasseneinteilung sind diese Klasseneinteilungen
für eine Analyse innerhalb des Bildes nicht möglich, da hier in einem Bild alle möglichen Facetten eines Einflussfaktors dargestellt werden. Daher werden die
Analysen des zweiten Auswerteschritts mit einer Clusternalyse durchgeführt. Der Vorteil der Clusteranalyse besteht darin, das keine Klassifikation vorgegeben
werden muss.
Als Beispiel wird das Falschem Mehltau, (Plasmopara viticola))
Versuchen gewählt1. Parallel zu den Blattmessungen mit der Plant
Probe wurden ganze Blätter mit einer hyperspektralen Linienkamera gescannt. Das RGB
Bild (rechts) ist ein Ausschnitt der Oberseite eines Blattes. Die Frage gilt zu klären
was, welche pathogen bedingten Muster der Blattunterseite durch die
Modellanpassung auf der Blattoberseite erkannt werden können. Dazu wird das Problem nach der Modellanpassung in mehrere Teilschritte
aufgeteilt.
Teil A: Bildhafte Darstellung der Modellparameter (Darstellung in 8 äquidistanten Farbklassen) und Clusteranalyse


Die Modellparameter, die die relative Reflexion auf der y-Achse beschreiben, ergeben schon ein sehr gutes Bild der Vorlage (Links), einige Parameter sind physiologisch wichtig, wie der Wendepunkt in den Infrarotbereich (Mitte), andere Parameter des Modells besitzen nur eine schwache visuelle Übereinstimmung mit der RGB Vorlage.
Die Clusteranalyse über alle Modellparameter ergab je nach Anzahl der Vorgaben (von links 3, 5, 8 Cluster) folgende Muster:



Ergebnis: Mit drei Clustern (Bild links) lassen sich die Bereiche "Gesund" (befallsfrei, grüner Bereich), "krank" (befallen, Ocker) und "nekrotisches Gewebe" (tot, gelb) unterscheiden; bei 5 bzw. 8 Clustern werden die Klassen feiner unterteilt, was visuell nicht nur zu keinem Vorteil führt, sondern eine Re-Identifizierung des Geschehens tatsächlich erschwert. Hinzu kommt, dass die Cluster sich mit anderen Datensätzen nicht so leicht vergleichen lassen, und die Cluster nicht linear angeordnet sind.
Teil B: Identifizierung von empirischen Maßzahlen zur Beschreibung und Klassifizierung des Pathogendrucks.
Basierend auf der Idee, dass einige Modellparameter die Bildsituation sehr gut beschreiben, wurde die Hypothese getestet, ob diese Parameter auf der Basis entsprechender Indices zusammengefasst werden können. Die einfachste Möglichkeit sind Summen, aber das Verfahren ist offen für beliebige Kombinationen.
Die interessanten Parameter werden zu unterschiedlichen empirischen dimensionslosen Maßzahlen unterschiedlicher Komplexität zusammengefasst. Die Klassentrennung erfolgt über eine Clusteranalyse des Trainingsdatensatzes. Getestet werden 2 bis 8 Cluster, d.h. die Gesamtstreuung wird entsprechend der vorgegebenen Klassen unterteilt. Die Anzahl der Cluster ist willkürlich gewählt worden, eine noch höhere Auflösung bringt keinen weiteren Vorteil. Die Clusteranalyse für jede Maßzahl beinhaltet 2 Vorteile: Die Cluster lassen sich leicht der Maßzahl nach sortieren und die Abbildungen über die Cluster sind bei einer vorgegebenen Farbskala direkt vergleichbar, was wichtig für die relative Beurteilung eines Verfahrens ist. Je nach Maßzahl ergeben sich unterschiedliche Klassenbereiche, die nur vereinzelt äquidistant sind.
Plausibilitätsprüfung der Maßzahlen
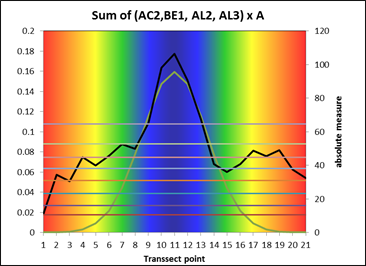
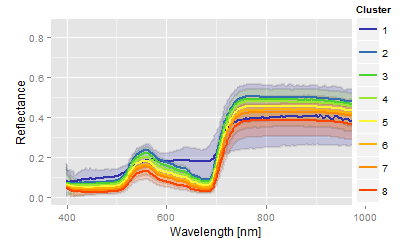
Zur Überprüfung dieser Maßzahlen wurde aus dem Vorlagebild ein Transsektbereich gewählt, der das ganze Spektrum von "Gesund" über "Krank" bis "Nekrose" und wieder zurück umfasst. Dieser Transsekt wird in den entsprechenden Spektralfarben von rot = Gesund bis blau = Nekrose mit den relativen Übergängen im gelb-grün Bereich dargestellt. Anbei ein paar Beispiele für 8 vorgegebene Cluster:
| Maßzahl | Transsekt und Verteilung der 8 Klassen, Maßzahlen werden auf der rechten y-Achse dargestellt | Aus Platzgründen werden nur die Extreme dargestellt |
|---|---|---|
| Erläuterung | R² zu gesund, d.h. es werden die Abweichungsquadrate zu einem gesunden Spektrum berechnet, so etwas wie eine Standardmethode, falls eine obere Grenze definiert werden kann. Rotbereich: gesund; blau: krankes Gewebe | Maßzahl aus der Summe einiger Parameter mal einem Höhenparameter. |
| Transsekt und Verteilung der 8 Klassen, Maßzahlen werden auf der rechten y-Achse dargestellt | 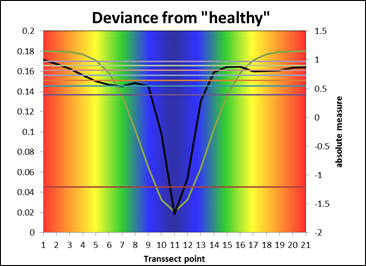 |
 |
| Mediane Spektralverläufe der 8 Cluster inkl. der 95% Quantile | 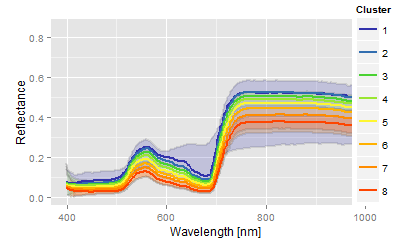 |
 |
| resultierendes Hyperspektralbild in Clusterfarben (Spektralschema), rot: gesundes Gewebe, blau: befallenes und nekrotisches Gewebe |  |
 |
| Resümee | Klare Abgrenzung der Informationen, der Übergang in den Stressbereich wird zu spät erkannt, gewisse Subjektivität durch Definition eines „gesunden“ Spektrums. Potentiell guter Ansatz, schwach im Übergang zu stärkerem Stress. | Auch hier eine klare Abgrenzung der Klassen, der Übergang in den Stressbereich ist plausibel, die Reaktion auf Nekrosen intensiv. Potentiell ein Ansatz, der die beste Beschreibung für krankheitsbedingte Übergänge liefert. |
 Evaluation reinvented
Evaluation reinvented